Algumas pessoas têm questionado sobre os resultados de Análise de Similaridade (ANOSIM) fornecidos através do pacote "vegan" na plataforma do R. Os resultados providos apenas mostram o R global, relativo à estatística R da análise, e o p-valor, evidenciando unicamente se há diferença florística entre as comunidades, mas não mostram os p-valores entre todos os pares das comunidades em questão.
Exemplo de output da ANOSIM do "vegan":
Mas então, qual a outra a opção para verificar entre quais pares de comunidades as diferenças florísticas são significativas?
A opção mais prática e de fácil acesso e uso é conduzir uma ANOSIM-One Way no software Past. O link abaixo mostra rapidamente e passo a passo como fazer isso no Past.
Você irá inserir no Past a mesma matriz de espécies utilizada para a análise no "vegan". Na barra lateral esquerda cinza do Past você deverá inserir os nomes das comunidades correspondentes as espécies nas colunas na quantidade correspondente para cada comunidade. Ou seja, se as 50 primeiras linhas correspondem à comunidade A, então marque com alguma cor (ex: vermelho, azul...) essas 50 linhas identificando tal comunidade (ex: Comun.A). O Past não roda a análise se as linhas pertencentes a cada comunidade não forem discriminadas em cores distintas. Após inserir os dados da matriz de espécies e discriminar suas comunidades nas linhas como mencionado, basta selecionar tudo e ir em "Multivar" -> "Anosim-One Way".
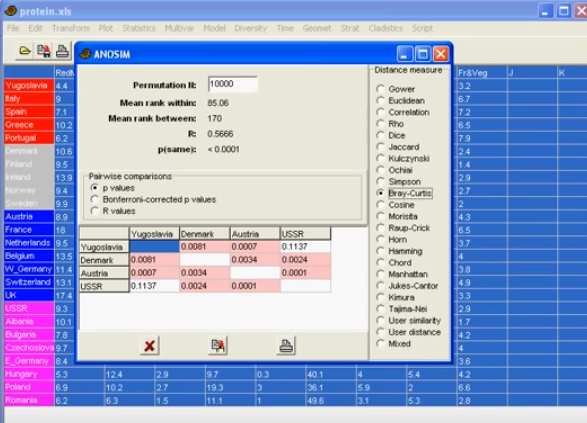
Exemplo de output de ANOSIM-One Way no Past, mostrando os p-valores de comparações aos pares entre comunidades:
Outra coisa fundamental a se considerar antes de rodar sua ANOSIM é a distância de dissimilaridade que você escolhe. É comumente aceito que para matrizes de espécies baseadas em presença e ausência usa-se distância de Jaccard e para matrizes baseadas em abundância usa-se Bray-Curtis.
Links relacionados:
Diferenças e afinidades entre Jaccard e Bray-Curtis:
Manual do Past em Português:
Manual original em Inglês: